如果说 K-means 和 GMM 这些聚类的方法是古代流行的算法的话,那么这次要讲的 Spectral Clustering 就可以算是现代流行的算法了,中文通常称为“谱聚类”。由于使用的矩阵的细微差别,谱聚类实际上可以说是一“类”算法。
Spectral Clustering 和传统的聚类方法(例如 K-means)比起来有不少优点:
- 和 K-medoids 类似,Spectral Clustering 只需要数据之间的相似度矩阵就可以了,而不必像 K-means 那样要求数据必须是 N 维欧氏空间中的向量。
- 由于抓住了主要矛盾,忽略了次要的东西,因此比传统的聚类算法更加健壮一些,对于不规则的误差数据不是那么敏感,而且 performance 也要好一些。许多实验都证明了这一点。事实上,在各种现代聚类算法的比较中,K-means 通常都是作为 baseline 而存在的。
- 计算复杂度比 K-means 要小,特别是在像文本数据或者平凡的图像数据这样维度非常高的数据上运行的时候。
突然冒出这么一个要求比 K-means 要少,结果比 K-means 要好,算得还比 K-means 快的东西,实在是让人不得不怀疑是不是江湖骗子啊。所以,是骡子是马,先拉出来溜溜再说。不过,在 K-medoids 那篇文章中曾经实际跑过 K-medoids 算法,最后的结果也就是一个 accuracy ,一个数字又不能画成图表之类的,看起来实在是没意思,而且 K-means 跑起来实在是太慢了,所以这里我还是稍微偷懒一下,直接引用一下一篇论文里的结果吧。
结果来自论文 Document clustering using locality preserving indexing 这篇论文,这篇论文实际上是提的另一种聚类方法(下次如果有机会也会讲到),不过在它的实验中也有 K-means 和 Spectral Clustering 这两组数据,抽取出来如下所示:
| k | TDT2 | Reuters-21578 | ||
| K-means | SC | K-means | SC | |
| 2 | 0.989 | 0.998 | 0.871 | 0.923 |
| 3 | 0.974 | 0.996 | 0.775 | 0.816 |
| 4 | 0.959 | 0.996 | 0.732 | 0.793 |
| … | ||||
| 9 | 0.852 | 0.984 | 0.553 | 0.625 |
| 10 | 0.835 | 0.979 | 0.545 | 0.615 |
1 | 1 |
其中 TDT2 和 Reuters-21578 分别是两个被广泛使用的标准文本数据集,虽然在不同的数据集上得出来的结果并不能直接拿来比较,但是在同一数据集上 K-means 和 SC (Spectral Clustering) 的结果对比就一目了然了。实验中分别抽取了这两个数据集中若干个类别(从 2 类到 10 类)的数据进行聚类,得出的 accuracy 分别在上表中列出(我偷懒没有全部列出来)。可以看到,Spectral Clustering 这里完胜 K-means 。
这么强大的算法,再戴上“谱聚类”这么一个高深莫测的名号,若不是模型无比复杂、包罗宇宙,就肯定是某镇山之宝、不传秘籍吧?其实不是这样,Spectral Clustering 不管从模型上还是从实现上都并不复杂,只需要能求矩阵的特征值和特征向量即可──而这是一个非常基本的运算,任何一个号称提供线性代数运算支持的库都理应有这样的功能。而关于 Spectral Clustering 的秘籍更是满街都是,随便从地摊上找来一本,翻开便可以看到 Spectral Clustering 算法的全貌:
- 根据数据构造一个 Graph ,Graph 的每一个节点对应一个数据点,将相似的点连接起来,并且边的权重用于表示数据之间的相似度。把这个 Graph 用邻接矩阵的形式表示出来,记为
 。一个最偷懒的办法就是:直接用我们前面在 K-medoids 中用的相似度矩阵作为
。一个最偷懒的办法就是:直接用我们前面在 K-medoids 中用的相似度矩阵作为  。
。 - 把
 的每一列元素加起来得到
的每一列元素加起来得到  个数,把它们放在对角线上(其他地方都是零),组成一个
个数,把它们放在对角线上(其他地方都是零),组成一个  的矩阵,记为
的矩阵,记为  。并令
。并令  。
。 - 求出
 的前
的前  个特征值(在本文中,除非特殊说明,否则“前
个特征值(在本文中,除非特殊说明,否则“前  个”指按照特征值的大小从小到大的顺序)
个”指按照特征值的大小从小到大的顺序) 以及对应的特征向量
以及对应的特征向量  。
。 - 把这
 个特征(列)向量排列在一起组成一个
个特征(列)向量排列在一起组成一个  的矩阵,将其中每一行看作
的矩阵,将其中每一行看作  维空间中的一个向量,并使用 K-means 算法进行聚类。聚类的结果中每一行所属的类别就是原来 Graph 中的节点亦即最初的
维空间中的一个向量,并使用 K-means 算法进行聚类。聚类的结果中每一行所属的类别就是原来 Graph 中的节点亦即最初的 个数据点分别所属的类别。
个数据点分别所属的类别。
就是这么几步,把数据做了一些诡异的变换,然后还在背后偷偷地调用了 K-means 。到此为止,你已经可以拿着它上街去招摇撞骗了。不过,如果你还是觉得不太靠谱的话,不妨再接着往下看,我们再来聊一聊 Spectral Clustering 那几个“诡异变换”背后的道理何在。
其实,如果你熟悉 Dimensional Reduction (降维)的话,大概已经看出来了,Spectral Clustering 其实就是通过 Laplacian Eigenmap 的降维方式降维之后再做 K-means 的一个过程──听起来土多了。不过,为什么要刚好降到 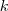 维呢?其实整个模型还可以从另一个角度导出来,所以,让我们不妨先跑一下题。
维呢?其实整个模型还可以从另一个角度导出来,所以,让我们不妨先跑一下题。
在 Image Processing (我好像之前有听说我对这个领域深恶痛绝?)里有一个问题就是对图像进行Segmentation (区域分割),也就是让相似的像素组成一个区域,比如,我们一般希望一张照片里面的人(前景)和背景被分割到不同的区域中。在 Image Processing 领域里已经有许多自动或半自动的算法来解决这个问题,并且有不少方法和 Clustering 有密切联系。比如我们在谈 Vector Quantization 的时候就曾经用 K-means 来把颜色相似的像素聚类到一起,不过那还不是真正的 Segmentation ,因为如果仅仅是考虑颜色相似的话,图片上位置离得很远的像素也有可能被聚到同一类中,我们通常并不会把这样一些“游离”的像素构成的东西称为一个“区域”,但这个问题其实也很好解决:只要在聚类用的 feature 中加入位置信息(例如,原来是使用 R、G、B 三个值来表示一个像素,现在加入 x、y 两个新的值)即可。
另一方面,还有一个经常被研究的问题就是 Graph Cut ,简单地说就是把一个 Graph 的一些边切断,让他被打散成一些独立联通的 sub-Graph ,而这些被切断的边的权值的总和就被称为 Cut 值。如果用一张图片中的所有像素来组成一个 Graph ,并把(比如,颜色和位置上)相似的节点连接起来,边上的权值表示相似程度,那么把图片分割为几个区域的问题实际上等价于把 Graph 分割为几个 sub-Graph 的问题,并且我们可以要求分割所得的 Cut 值最小,亦即:那些被切断的边的权值之和最小,直观上我们可以知道,权重比较大的边没有被切断,表示比较相似的点被保留在了同一个 sub-Graph 中,而彼此之间联系不大的点则被分割开来。我们可以认为这样一种分割方式是比较好的。
实际上,抛开图像分割的问题不谈,在 Graph Cut 相关的一系列问题中,Minimum cut (最小割)本身就是一个被广泛研究的问题,并且有成熟的算法来求解。只是单纯的最小割在这里通常并不是特别适用,很多时候只是简单地把和其他像素联系最弱的那一个像素给分割出去了,相反,我们通常更希望分割出来的区域(的大小)要相对均匀一些,而不是一些很大的区块和一些几乎是孤立的点。为此,又有许多替代的算法提出来,如 Ratio Cut 、Normalized Cut 等。
不过,在继续讨论之前,我们还是先来定义一下符号,因为仅凭文字还是很难表述清楚。将 Graph 表示为邻接矩阵的形式,记为 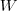 ,其中
,其中 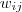 是节点
是节点  到节点
到节点 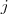 的权值,如果两个节点不是相连的,权值为零。设
的权值,如果两个节点不是相连的,权值为零。设 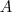 和
和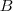 为 Graph 的两个子集(没有交集),那么两者之间的 cut 可以正式定义为:
为 Graph 的两个子集(没有交集),那么两者之间的 cut 可以正式定义为:
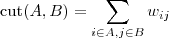
先考虑最简单的情况,如果把一个 Graph 分割为两个部分的话,那么 Minimum cut 就是要最小化 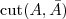 (其中
(其中 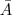 表示
表示 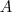 的子集)。但是由于这样经常会出现孤立节点被分割出来的情况,因此又出现了 RatioCut :
的子集)。但是由于这样经常会出现孤立节点被分割出来的情况,因此又出现了 RatioCut :
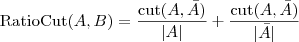
以及 NormalizedCut :
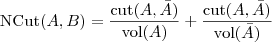
其中  表示
表示 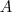 中的节点数目,而
中的节点数目,而  。两者都可以算作
。两者都可以算作 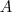 的“大小”的一种度量,通过在分母上放置这样的项,就可以有效地防止孤立点的情况出现,达到相对平均一些的分割。事实上,Jianbo Shi 的这篇 PAMI paper:Normalized Cuts and Image Segmentation 正是把 NormalizedCut 用在图像分割上了。
的“大小”的一种度量,通过在分母上放置这样的项,就可以有效地防止孤立点的情况出现,达到相对平均一些的分割。事实上,Jianbo Shi 的这篇 PAMI paper:Normalized Cuts and Image Segmentation 正是把 NormalizedCut 用在图像分割上了。
搬出 RatioCut 和 NormalizedCut 是因为它们和这里的 Spectral Clustering 实际上有非常紧密的联系。看看 RatioCut ,式子虽然简单,但是要最小化它却是一个 NP 难问题,不方便求解,为了找到解决办法,让我们先来做做变形。
令  表示 Graph 的所有节点的集合,首先定义一个
表示 Graph 的所有节点的集合,首先定义一个 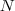 维向量
维向量  :
:

再回忆一下我们最开始定义的矩阵 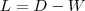 ,其实它有一个名字,叫做 Graph Laplacian ,不过,我们后面可以看到,其实有好几个类似的矩阵都叫做这个名字:
,其实它有一个名字,叫做 Graph Laplacian ,不过,我们后面可以看到,其实有好几个类似的矩阵都叫做这个名字:
Usually, every author just calls “his” matrix the graph Laplacian.
其实也可以理解,就好象现在所有的厂家都说自己的技术是“云计算”一样。这个 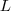 有一个性质就是:
有一个性质就是:

这个是对任意向量  都成立的,很好证明,只要按照定义展开就可以得到了。把我们刚才定义的那个
都成立的,很好证明,只要按照定义展开就可以得到了。把我们刚才定义的那个  带进去,就可以得到
带进去,就可以得到

另外,如果令  为各个元素全为 1 的向量的话,直接展开可以很容易得到
为各个元素全为 1 的向量的话,直接展开可以很容易得到  和
和  。由于
。由于  是一个常量,因此最小化 RatioCut 就等价于最小化
是一个常量,因此最小化 RatioCut 就等价于最小化  ,当然,要记得加上附加条件
,当然,要记得加上附加条件  以及
以及  。
。
问题转化到这个样子就好求了,因为有一个叫做 Rayleigh quotient 的东西:

他的最大值和最小值分别等于矩阵 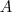 的最大的那个特征值和最小的那个特征值,并且极值在
的最大的那个特征值和最小的那个特征值,并且极值在  等于对应的特征向量时取到。由于
等于对应的特征向量时取到。由于  是常数,因此最小化
是常数,因此最小化  实际上也就等价于最小化
实际上也就等价于最小化  ,不过由于
,不过由于 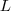 的最小的特征值为零,并且对应的特征向量正好为
的最小的特征值为零,并且对应的特征向量正好为  (我们这里仅考虑 Graph 是联通的情况),不满足
(我们这里仅考虑 Graph 是联通的情况),不满足  的条件,因此我们取第二个小的特征值,以及对应的特征向量
的条件,因此我们取第二个小的特征值,以及对应的特征向量  。
。
到这一步,我们看起来好像是很容易地解决了前面那个 NP 难问题,实际上是我们耍了一个把戏:之前的问题之所以 NP 难是因为向量  的元素只能取两个值
的元素只能取两个值  和
和  中的一个,是一个离散的问题,而我们求的的特征向量
中的一个,是一个离散的问题,而我们求的的特征向量  其中的元素可以是任意实数,就是说我们将原来的问题限制放宽了。那如何得到原来的解呢?一个最简单的办法就是看
其中的元素可以是任意实数,就是说我们将原来的问题限制放宽了。那如何得到原来的解呢?一个最简单的办法就是看  的每个元素是大于零还是小于零,将他们分别对应到离散情况的
的每个元素是大于零还是小于零,将他们分别对应到离散情况的  和
和  ,不过我们也可以采取稍微复杂一点的办法,用
,不过我们也可以采取稍微复杂一点的办法,用  的 K-means 来将
的 K-means 来将  的元素聚为两类。
的元素聚为两类。
到此为止,已经有 Spectral Clustering 的影子了:求特征值,再对特征向量进行 K-means 聚类。实际上,从两类的问题推广到 k 类的问题(数学推导我就不再详细写了),我们就得到了和之前的 Spectral Clustering 一模一样的步骤:求特征值并取前 k 个最小的,将对应的特征向量排列起来,再按行进行 K-means 聚类。分毫不差!
用类似的办法,NormalizedCut 也可以等价到 Spectral Clustering 不过这次我就不再讲那么多了,感兴趣的话(还包括其他一些形式的 Graph Laplacian 以及 Spectral Clustering 和 Random walk 的关系),可以去看这篇 Tutorial :A Tutorial on Spectral Clustering 。
为了缓和一下气氛,我决定贴一下 Spectral Clustering 的一个简单的 Matlab 实现:
2
2
最后,我们再来看一下本文一开始说的 Spectral Clustering 的几个优点:
- 只需要数据的相似度矩阵就可以了。这个是显然的,因为 Spectral Clustering 所需要的所有信息都包含在
 中。不过一般
中。不过一般  并不总是等于最初的相似度矩阵——回忆一下,
并不总是等于最初的相似度矩阵——回忆一下, 是我们构造出来的 Graph 的邻接矩阵表示,通常我们在构造 Graph 的时候为了方便进行聚类,更加强到“局部”的连通性,亦即主要考虑把相似的点连接在一起,比如,我们设置一个阈值,如果两个点的相似度小于这个阈值,就把他们看作是不连接的。另一种构造 Graph 的方法是将 n 个与节点最相似的点与其连接起来。
是我们构造出来的 Graph 的邻接矩阵表示,通常我们在构造 Graph 的时候为了方便进行聚类,更加强到“局部”的连通性,亦即主要考虑把相似的点连接在一起,比如,我们设置一个阈值,如果两个点的相似度小于这个阈值,就把他们看作是不连接的。另一种构造 Graph 的方法是将 n 个与节点最相似的点与其连接起来。 - 抓住了主要矛盾,忽略了次要的东西,Performance 比传统的 K-means 要好。实际上 Spectral Clustering 是在用特征向量的元素来表示原来的数据,并在这种“更好的表示形式”上进行 K-means 。实际上这种“更好的表示形式”是用 Laplacian Eigenmap 进行降维的后的结果,如果有机会,下次谈 Dimensionality Reduction 的时候会详细讲到。而降维的目的正是“抓住主要矛盾,忽略次要的东西”。
- 计算复杂度比 K-means 要小。这个在高维数据上表现尤为明显。例如文本数据,通常排列起来是维度非常高(比如,几千或者几万)的稀疏矩阵,对稀疏矩阵求特征值和特征向量有很高效的办法,得到的结果是一些 k 维的向量(通常 k 不会很大),在这些低维的数据上做 K-means 运算量非常小。但是对于原始数据直接做 K-means 的话,虽然最初的数据是稀疏矩阵,但是 K-means 中有一个求 Centroid 的运算,就是求一个平均值:许多稀疏的向量的平均值求出来并不一定还是稀疏向量,事实上,在文本数据里,很多情况下求出来的 Centroid 向量是非常稠密,这时再计算向量之间的距离的时候,运算量就变得非常大,直接导致普通的 K-means 巨慢无比,而 Spectral Clustering 等工序更多的算法则迅速得多的结果。
说了这么多,好像有些乱,不过也只能到此打住了。最后再多嘴一句,Spectral Clustering 名字来源于Spectral theory ,也就是用特征分解来分析问题的理论了。
=================================================================
什么叫Spectral Algorithm?
广义上来说,任何在演算法中用到SVD/特征值分解的,都叫Spectral Algorithm。 从很老很老的PCA/LDA,到比较近的Spectral Embedding/Clustering,都属于这类。
为什么要用SVD/特征值分解?
其实并不是为用而用,而是不得不用。 目前在研究领域碰到的很多基础问题都是NP-hard的,找一个比较好的近似演算法要费很大的精力;就算找到多项式的近似方法,也会出现实际使用上仍然太慢/解陷入局部极小等问题。
比如说用K-means聚类,建模本身已经够简单了,但它是NP-hard的,用传统的EM迭代作近似解会陷入局部极小。
反之,SVD理论上只有唯一解,演算法速度相对又快,并且有大量理论结果及周边性质支持,可以算是一个很理想地能将NP-hard问题“靠”上去的模型;它的另一个好处是,作为带约束二次规划的一种特殊情况,它对运算式为二次的目标函数的“相容性”比较好,“靠”所要求的数学技巧不高,任何人,任何方向都能拿来试试。
Spectral Algorithm的几个方向:
传统的如PCA/LDA用来做线性降维,2000年左右的一些Spectral Embedding及
Spectral Clustering,还有周边的一些,如Low-rank approximation等等。
Spectral Clustering,中文通常称为“谱聚类”。由于使用的矩阵的细微差别,谱聚类实际上可以说是一“类”算法。
Spectral Clustering 和传统的聚类方法(例如 K-means)比起来有不少优点:
1)和 K-medoids 类似,Spectral Clustering 只需要数据之间的相似度矩阵就可以了,而不必像 K-means 那样要求数据必须是 N 维欧氏空间中的向量。
2)由于抓住了主要矛盾,忽略了次要的东西,因此比传统的聚类算法更加健壮一些,对于不规则的误差数据不是那么敏感,而且 performance 也要好一些。许多实验都证明了这一点。事实上,在各种现代聚类算法的比较中,K-means 通常都是作为 baseline 而存在的。
3)计算复杂度比 K-means 要小,特别是在像文本数据或者平凡的图像数据这样维度非常高的数据上运行的时候。
Spectral Clustering 算法的全貌:
1)根据数据构造一个 Graph ,Graph 的每一个节点对应一个数据点,将相似的点连接起来,并且边的权重用于表示数据之间的相似度。把这个 Graph 用邻接矩阵的形式表示出来,记为 W 。
2)把每一列元素加起来得到N 个数,把它们放在对角线上(其他地方都是零),组成一个N*N的矩阵,记为D 。并令L = D – W 。
3)求出L的前k个特征值(在本文中,除非特殊说明,否则“前k个”指按照特征值的大小从小到大的顺序)以及对应的特征向量。
4)把这k个特征(列)向量排列在一起组成一个N*k的矩阵,将其中每一行看作k维空间中的一个向量,并使用 K-means 算法进行聚类。聚类的结果中每一行所属的类别就是原来 Graph 中的节点亦即最初的N个数据点分别所属的类别。
下面是Spectral Clustering 的一个简单的 Matlab 实现:
function idx = spectral_clustering(W, k)
D = diag(sum(W));
L = D-W;
opt = struct('issym', true, 'isreal', true);
[V dummy] = eigs(L, D, k, 'SM', opt);
idx = kmeans(V, k);
end
最后,我们再来看一下本文一开始说的 Spectral Clustering 的几个优点:
1)只需要数据的相似度矩阵就可以了。这个是显然的,因为 Spectral Clustering 所需要的所有信息都包含在W中。不过一般W并不总是等于最初的相似度矩阵——回忆一下, 是我们构造出来的 Graph 的邻接矩阵表示,通常我们在构造 Graph 的时候为了方便进行聚类,更加强到“局部”的连通性,亦即主要考虑把相似的点连接在一起,比如,我们设置一个阈值,如果两个点的相似度小于这个阈值,就把他们看作是不连接的。另一种构造 Graph 的方法是将 n 个与节点最相似的点与其连接起来。
2)抓住了主要矛盾,忽略了次要的东西,Performance 比传统的 K-means 要好。实际上 Spectral Clustering 是在用特征向量的元素来表示原来的数据,并在这种“更好的表示形式”上进行 K-means 。
3)计算复杂度比 K-means 要小。这个在高维数据上表现尤为明显。例如文本数据,通常排列起来是维度非常高(比如,几千或者几万)的稀疏矩阵,对稀疏矩阵求特征值和特征向量有很高效的办法,得到的结果是一些 k 维的向量(通常 k 不会很大),在这些低维的数据上做 K-means 运算量非常小。但是对于原始数据直接做 K-means 的话,虽然最初的数据是稀疏矩阵,但是 K-means 中有一个求 Centroid 的运算,就是求一个平均值:许多稀疏的向量的平均值求出来并不一定还是稀疏向量,事实上,在文本数据里,很多情况下求出来的 Centroid 向量是非常稠密,这时再计算向量之间的距离的时候,运算量就变得非常大,直接导致普通的 K-means 巨慢无比,而 Spectral Clustering 等工序更多的算法则迅速得多的结果。
作者:洞庭散人
出处:http://phinecos.cnblogs.com/
为什么先做降维再做K-means,效果会更好呢?
另外,有趣的是K-means可以用PCA来做近似解。 K-means是说找到K个点,使得所有点到这K个点的距离平方和最小;
而SVD是说找到一个子空间,使得所有点到这个子空间的距离平方和最小。 于是这两者就建立了联系,K-means便relax到SVD上去了。
Spectral Clustering/Embedding:
Spectral Clustering可算是Spectral Algorithm的重头戏。
所谓Clustering,就是说聚类,把一堆东西(合理地)分成两份或者K份。 从数学上来说,聚类的问题就相当于Graph Partition的问题,即给定一个图G = (V, E),如何把它的顶点集划分为不相交的子集,使得这种划分最好。 其难点主要有两个:
1.这个“合理”其实相当难达到,随便设一个目标函数可能达不到希望的结果。 大家可以看了看[1] Ravi Kannan and Adrian Vetta, On clusterings: good, bad and spectral,这里详细地讨论了一下准则的选择问题。
2.即使我们定义了一个相当好的聚类准则,如何优化它又是一个问题。
对于1,在
Spectral Clustering这一块,各家有各家的想法。 主要有以下几种:
a)大名鼎鼎的Normalized Cut[2],还有一些变种如Ratio Cut/Minmax cut.
b)和代数图论紧密相联的Minimum conductance[1].
c)没有准则,但有证明的演算法[3]
d)不基于图,而是reformulate原来的聚类方法,使之变成SVD能解的问题[4]。
2则完全被1的选取所决定。
Normalized Cut:
在图上,定义什么样的聚类最好,最简单的方法是圈定K个不相交顶点集之后,希望顶点集之间的边,其权值的和最小。 (边上的权值代表的是两头的顶点邻近的程度,或者说相似度)这就是所谓MinCut(最小割)问题。 二类分类的最小割不是NP-hard的,但是这不能让人感到开心,因为MinCut这个准则对于聚类不好。
具体来说,Mincut完全可能将离大部队过远的单个顶点与其他顶点分开,形成两类。
事实上,我们不仅仅要让割边的权和最小,而且要让这K个顶点集都差不多大,这样才符合聚类给人的直观感觉。
于是在MinCut的基础上,出现了Normalized Cut.思路很简单,将Cut normalize一下,除以表现顶点集大小的某种量度(如vol A =所有A中顶点集的度之和)。
也就是Normalize Cut(A, B) = Cut(A, B) / volA + cut(A, B) / volB
然而这样一改,NP-hard就来了。 这几乎是所有组合优化问题的恶梦。
怎么办呢? 把组合优化问题连续化,即所谓减少约束,进行适当的relax。 那么为什么会和SVD扯上的呢?
很简单,聚类是东西分成不相交集,也就是有正交的含义在里面;只是分东西必须是0-1式的,这种离散化,就是np-hard的原因。 我们把正交约束保留,但把离散变成连续的,聚类就变成了寻找(列)正交阵的优化问题,那正是SVD的火力所在!
就这样,通过这种巧妙的relax,NP-hard问题有了近似解。 且不说这近似解的质量如何,这种方法是相当令人振奋的。 (关于Normalized Cut近似解的质量,似乎没有什么文章能够给出严格的证明,只是实际效果不错就是了。)
值得一提的是,Normalized Cut还和图上的Markov chain有紧密的关系[5]。 Normalized Cut这个量度,换成Markov chain的语言就是在图上随机游走,子集间相互“串门”的概率大小。 相当有趣。
 的矩阵,记为
的矩阵,记为 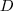 。并令
。并令 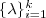 以及对应的特征向量
以及对应的特征向量 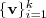 。
。 的矩阵,将其中每一行看作
的矩阵,将其中每一行看作 